微生态研究
研究正常微生物群的结构、功能及其与宿主相互依赖和相互制约关系的科学
项目介绍
元基因组学(Metagenomics),也称宏基因组学,研究以环境中所有微生物基因组为研究对象,通过对环境样品中的全基因组DNA进行高通量测序,获得单个样品的总的基因序列,在分析微生物多样性、种群结构、进化关系的基础上,可进一步探究微生物群体功能活性、相互协作关系及与环境之间的关系,发掘潜在的生物学意义。在人体微生态领域,元基因组技术的出现,更加清楚地揭示了微生物与人体之间的微妙关系,成功绕开了“不可培养微生物”对人体微生物深入研究的阻碍,为开发利用“不可培养微生物”基因组提供了坚实的技术支撑。目前,在人体微生物活性物质的开发与利用、微生物分布及功能动态变化分析、疾病和人体微生物的因果关系等方面有广泛应用。
生信分析
慕柏优势
~~~从实验设计到生信分析的一站式服务~~~
研究内容多样:通过对群落中所有微生物进行全基因组分析,研究群落的物种、基因、功能结构/差异,并可以和代谢组学等进行关联分析,深入研究微生物的作用机制。
可组装单菌:对于简单的环境,可以获得较好的单菌基因组,并进行深入的功能挖掘。
个性化分析:提供针对人体微生物的个性化分析/高级分析内容,其他个性化分析需求可与信息分析同事沟通。
结果展示
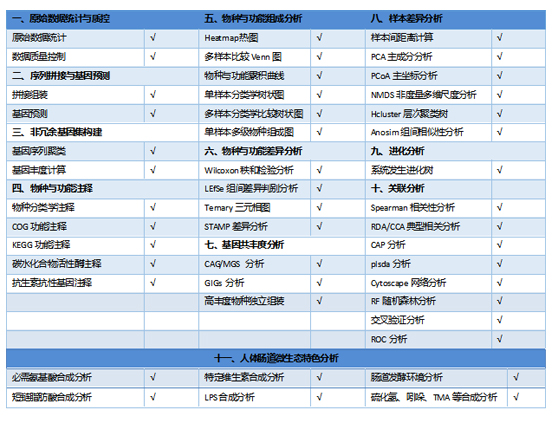
eggNOG分析:
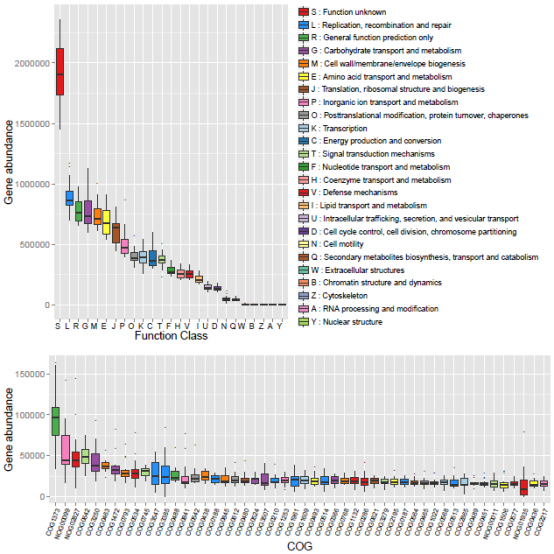
eggNOG (evolutionary genealogy of genes: Non-supervised Orthologous Groups,http://eggnog.embl.de/)数据库: 是国际上普遍认可的同源聚类基因群的专业注释数据库,使用BLASTP(BLAST Version 2.2.28+,http://blast.ncbi.nlm.nih.gov/Blast.cgi)将基因集序列与eggNOG数据库进行比对(BLAST比对参数设置期望值e-value为1e-5),获得基因对应的COG (Clusters of orthologous groups of proteins,直系同源序列聚类),然后使用COG对应的基因丰度总和计算该COG的丰度。
NetWork 网络图分析:
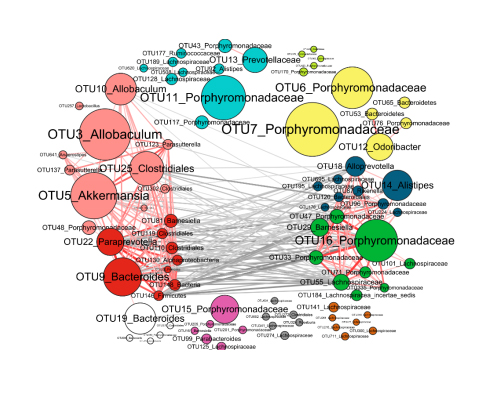
生态学中一般认为功能上关系密切的群落往往表现出丰度的“同升同降”,根据微生物群落丰度信息计算样本中物种之间的相关性,并据此划分不同的共变化组(Co-abundance group,CAG);可发现物种间的相互关系、CAG与生理指标或环境因子间的关系。
物种网络图中节点代表存在相互关系的物种,图中一个节点代表一个OTU,节点的大小代表了在样本中丰度的多少。同种颜色的节点代表一个CAG。节点间的连线的粗细代表OTU之间的相关性大小,线条越粗,相关性越大,图中只画出了相关性系数大于0.5的连线;红色线条表示正相关,灰色线条表示负相关。OTU共表达网络分析网络图的展示形式多变,一般显示样本间及物种间的关系,计算CAG时样本数须大于18个。


